嘻嘻嘻~~~,晚上秒变生信分析小白,一个游走在生物学和计算机变成之间的小白,享受着里面的快乐和痛苦。不停的挣扎,不停的成长,多学习,多尝试,一定会有意想不到的收获。加油!!!
首先,需要先说明的是本作图经验,完全模仿于果子学生信的一篇博文,所以先把链接丢在这里啦,自己写这篇博文,也完全是为了记录这个操作过程,好在下次需要的时候,更好的去回忆。话不多说,开始表演。
https://mp.weixin.qq.com/s?__biz=MzIyMzA2MTcwMg==&mid=2650733008&idx=1&sn=b66e3fd527f99ddf19dcf6c2501e5be3&chksm=f029aa79c75e236f8951b87e17a51dc6a7dfeb555b983d8dc6d5a8c756b3eacc21829f19dc86&token=1035043907&lang=zh_CN&scene=21#wechat_redirect
首先,做这个相关性分析,在这里需要安装几个R包。
BiocManager::install(c("tidyr","dplyr","ggstatsplot"))
##加载的数据,就是我们常见的TCGA的表达矩阵,只是需要先将其进行转置。
##我们在此用得是自己的一个Test的数据。其格式如下:
注意,在此处,基因名一定要作为列名,因为后面需用ggstatsplot进行散点图绘制的时候,其需要用到列名中的基因名字。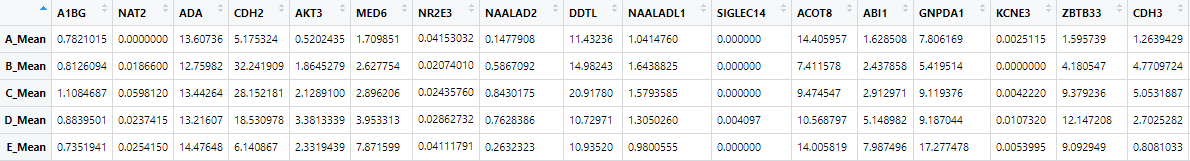
该数据,列名就是基因名,行名就是样本名。为了缩短数据处理时间,我们在此只取前500列作为后续的实验分析。
test3 <- test2[,1:500]
head(test3[1:3,1:3])
A1BG NAT2 ADA
A_Mean 0.7821015 0.000000 13.60736
B_Mean 0.8126094 0.018660 12.75982
C_Mean 1.1084687 0.059812 13.44264
y <- as.numeric(test3[,"A1BG"]) ## 本次操作中,选取A1BG基因作为需要分析的单基因,批量求取它与该表达矩阵中其他基因的相关性
colnames <- colnames(test3)
cor_data_df <- data.frame(colnames) ##构建这些基因的数据框
##利用 for循环来求A1BG与其他基因之间的相关性
library(tidyr)
library(dplyr)
for (i in 1:length(colnames))
test <- cor.test(as.numeric(test3[,i]),y,type="spearman")
cor_data_df[i,2] <- test$estimate
cor_data_df[i,3] <- test$p.value
names(cor_data_df) <- c("Symbol","correlation","pvalue")
head(cor_data_df)
Symbol correlation pvalue
1 A1BG 1.0000000 0.00000000
2 NAT2 0.8497555 0.06831151
3 ADA -0.3134327 0.60755911
4 CDH2 0.5979938 0.28680241
5 AKT3 0.2391356 0.69845042
6 MED6 -0.3618204 0.54957487
##提取p值小于0.05的数据
cor_data_sig <- cor_data_df %>%
filter(pvalue < 0.05) %>%
arrange(desc(abs(correlation)))%>%
dplyr::slice(1:500)
head(cor_data_sig)
Symbol correlation pvalue
1 A1BG 1.0000000 0.0000000000
2 NDUFC2-KCTD14 0.9920818 0.0008448014
3 C2CD4D 0.9761320 0.0044105842
4 LY75-CD302 0.9661582 0.0074352809
5 CEP295NL -0.9503865 0.0131666282
6 CORO7-PAM16 0.9455822 0.0151136100
library(ggstatsplot)
##利用该包进行绘制相关性的散点图和条形图
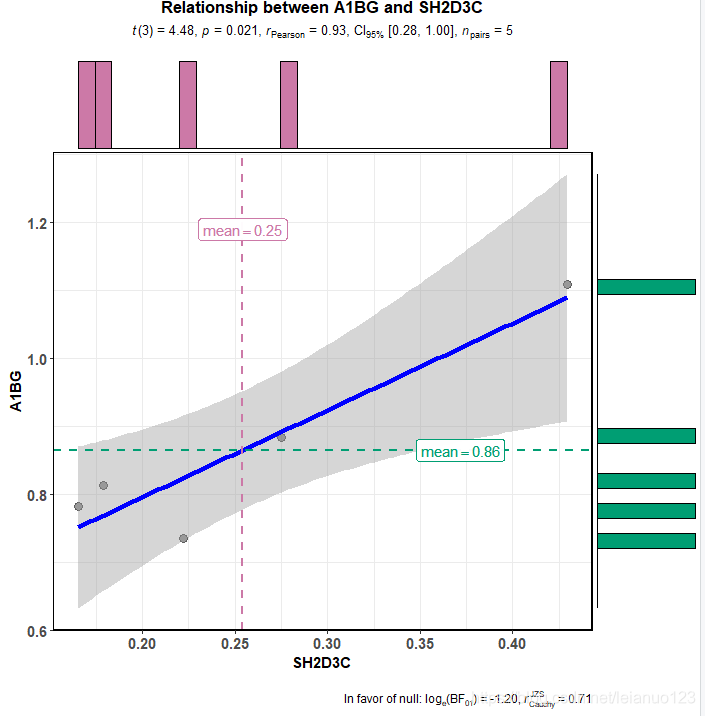
ggscatterstats(data =as.data.frame(test3),
y = A1BG,
x = SH2D3C,
centrality.para = "mean",
margins = "both",
xfill = "#CC79A7",
yfill = "#009E73",
marginal.type = "histogram",
title = "Relationship between A1BG and SH2D3C")
话不多说,上我的丑图。
其实大佬用的TCGA上的数据做出来的结果是这样的,比我的美多了,主要是别人的样本量较多,而我只有五个。

接着装另一个包,我们开始将多个图片拼接在一起。
BiocManager::install("cowplot")
library(cowplot)
dev.off() ##关掉上次的绘图
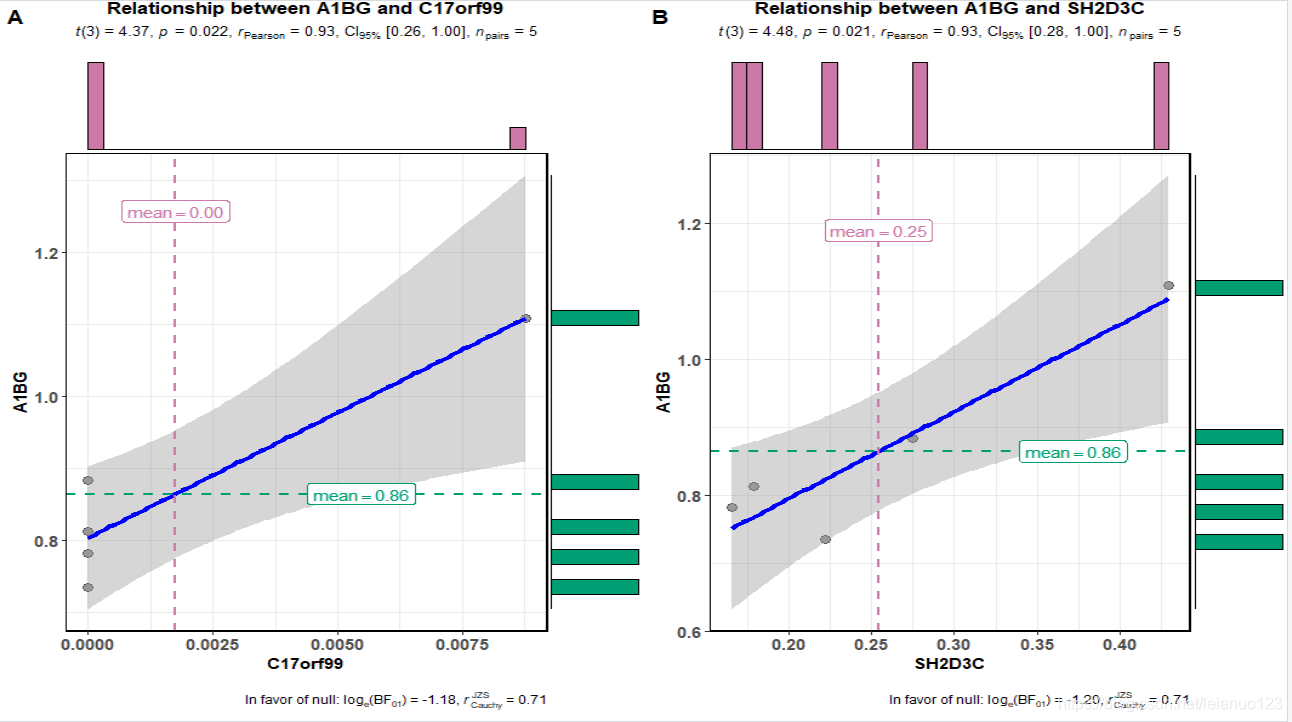
p1=ggscatterstats(data = as.data.frame(test3),
y = A1BG,
x = C17orf99,
centrality.para = "mean",
margins = "both",
xfill = "#CC79A7",
yfill = "#009E73",
marginal.type = "histogram",
title = "Relationship between A1BG and C17orf99")
P2=ggscatterstats(data =as.data.frame(test3),
y = A1BG,
x = SH2D3C,
centrality.para = "mean",
margins = "both",
xfill = "#CC79A7",
yfill = "#009E73",
marginal.type = "histogram",
title = "Relationship between A1BG and SH2D3C")
p2
2张图拼在一起的结果
接着进行GO分析
library(clusterProfiler)
#获得基因列表,说明由于我的测试数据质量不是特别好,因此取到的p<0.05的基因只有24条,在此,难以用于GO分析,所以我拿我的全部的基因来做GO分析
library(stringr)
gene <- str_trim(cor_data_sig$symbol,'both')##这是取出的相关性结果好的情况下
gene <- str_trim(Symbol,'both') ##这里Symbol是所有的基因名
#基因名称转换,返回的是数据框
gene = bitr(gene, fromType="SYMBOL", toType="ENTREZID", OrgDb="org.Hs.eg.db")
go <- enrichGO(gene = gene$ENTREZID, OrgDb = "org.Hs.eg.db", ont="all")
##将BP,CC,MF 三个GO类别按照ONTLOGY进行分类,将三个组分绘制在一张图上。
barplot(go, split="ONTOLOGY")+ facet_grid(ONTOLOGY~., scale="free")##条形图
dotplot(go, split="ONTOLOGY")+ facet_grid(ONTOLOGY~., scale="free")##气泡图
View(go)
在此献上丑图
GO的条形图
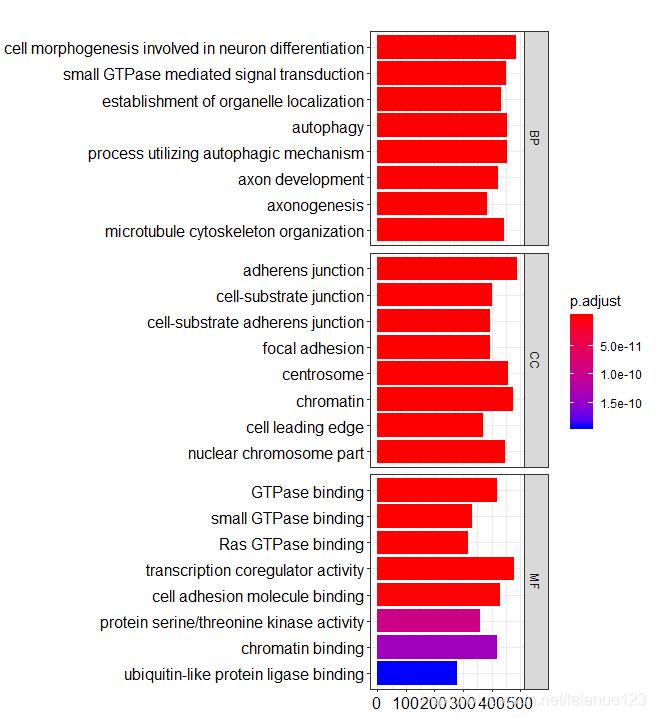
献上丑图,GO的气泡图
至此,此次的重复操作已经接近尾声了,图中的精彩和高潮部分,留给需要的人自己去体会,当然,后期的作图的精修,还需要根据自己的要求进行调整,第一次写R生信的博文,继续加油。
单基因批量相关性分析嘻嘻嘻~~~,晚上秒变生信分析小白,一个游走在生物学和计算机变成之间的小白,享受着里面的快乐和痛苦。不停的挣扎,不停的成长,多学习,多尝试,一定会有意想不到的收获。加油!!!首先,需要先说明的是本作图经验,完全模仿于果子学生信的一篇博文,所以先把链接丢在这里啦,自己写这篇博文,也完全是为了记录这个操作过程,好在下次需要的时候,更好的去回忆。话不多说,开始表演。https:...
TCGA|GEO可视化分析第1篇---相关性分析导读正文:
今天有小伙伴问我,筛选完差异基因后,想看自己关注的基因和其他基因的关系,应该怎么做?那当然要先做一下相关性分析了!好,下面让我给大家娓娓道来…
step1:我们先读取下所需要的数据
library(openxlsx)setwd("E:\\Bioinfo_analysis\\scripts\\corr\\corr_batch"...
TNBC-三阴性乳腺癌
三阴乳腺癌是指乳腺癌免疫组化结果:雌激素受体(ER)、孕激素受体(PR)和人表皮生长因子受体2(Her-2)均为阴性的乳腺癌。
三阴性乳腺癌容易早期发生广泛转移,如脑转移、骨转移等情况。所以,化疗在三阴性乳腺癌当中地位很重要。当然,复发高峰在手术以后头三年,过了复发高峰以后,预后也还是非常好的一种恶性肿瘤,具体要看分期如何、病人身体状况,以及接受什么样治疗等。
下载数据并提取
1.从TCGA.BRCA.sampleMap%2FBRCA
if __name__ == "__main__":
data = pd.read_csv("corpus-model-filtered.csv", header=None) #加载数据
data:pd.DataFrame = data.sample(frac=1.0)
数据可视化——R语言绘制散点相关图并自动添加相关系数和拟合方程加载所需的包并设置主题样式示例数据基本的散点相关图添加相关系数和显著性水平(P值)存在多个组别的散点相关图自动添加回归曲线的拟合方程拟合多项式方程
概述:通常,我们需要对两个连续变量进行相关性分析,可采用散点图叠加拟合曲线(有时也可包含95%置信区间)来呈现相关性分析的结果。本文将介绍使用R语言来绘制散点相关图,并自动添加相关系数和显著性水平(P值)及拟合方程。
使用工具: R语言中的ggplot2包、ggpubr包和ggpmisc包
本文大部分
忙里偷闲,在网上看到一绝美论文配图,制图优美,配色赏心悦目;是由作者IndrajeetPatil集合开发的ggstatsplot包,其实说白了,就是ggplot2的升级版,该升级版将箱线图与小提琴图进行了整合,话不多说,直接上操作!
组间的均数比较(ggbetweenstats函数)
#作物表型记录本#加载包library(ggstatsplot)library(ggplot2)#读取包自带数据,默认前6行head(movies_long)#使用ggbetweenstats函数进行参数设置并绘图ggb




