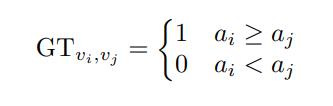[
0
.
0
1
,
0
.
0
0
5
,
0
.
0
0
1
,
0
.
0
0
0
5
,
0
.
0
0
0
1
]
4、损失函数:
-
Dice loss with or without squared prediction
-
Cross entropy loss
-
Cross entropy loss + Dice loss
-
Dice loss + Focal loss
二、网络配置编码
确定了搜索空间之后,就是将网络的这些候选配置进行编码,本文将所有的网络配置编码为一个一维向量
A
,对于每个块,可以使用5个整数进行表示,前三个数代表当前块的ID,块的类型,空间尺度,另外两个数表示与当前块相连的另外两个块的ID。其中第一个块(-1,-1),第二个块(0,-1)。
2、其他编码
本文按顺序应用
a
j
,则网络训练时的GT为
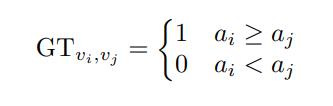
【在训练和测试的过程中,不同网络配置在验证集上得具体准确率并不重要,尤其是当图像数据不同的时候,所以该网络仅预测不同网络配置准确率之间的高低关系,这样也简化了网络训练的难度。】
四、训练过程
本文在LiTS-2017以及MSD数据集上进行实验。首先在搜索空间中随机选取100种网络配置,其中75种作为训练,25作为验证,然后对于每种网络配置训练迭代10000,则实际的训练数据量为75×75=5625【每两条向量作为一对进行输入】
五、实验结果
首先可以看一下搜索出来后最优的网络结构,如下图最右侧所示:

【实话实话,从结果来看这个网络结构比较”独特“,是否能够用在实际的工作中仍旧存疑】
其他的搜索结果:

这一部分可能对于各位同学实际工作的意义更大,具有一定的参考意义,实际使用的时候可以直接作为初始的设定,进行快速的模型训练。
实际的网络训练结果如下表所示:


不出意外的SOTA的结果,但是实际看上去和nnU-net的结果极为接近,文章在后面也做了和nn-Unet的对比,感兴趣的同学可以去看一下。
PyPM
ML
火花
PyPM
ML
-Spark是PySpark的Python PM
ML
评分库,称为Spark
ML
Transform
er
,它实际上是的Python API。
Java> = 1.8
Python 2.7或> = 3.5
PySpark
PySpark> = 3.0.0
PySpark> = 2.4.0,<3.0.0
pip install pypm
ml
-spark
或从github安装最新版本:
pip install --upgrade git+https://github.com/
auto
deployai/pypm
ml
-spark.git
整个网络是,先经过两层卷积,然后将featuremap切割成四份,分别经过四个并行的
transform
er
(heads可以自己设置),再将
上诉结果concat,再经过一个
transform
er
,然后就是一层层地decod
er
。
主要调试main.py、
transform
er
.py、build
er
s.py这三个文件就好,其余的只是依赖包
main.py:
运行文件。路径、数据集划分、以及测试和评价指标全在里头。
transform
er
:
这个文件里面包含了所有的网络模块(class)。
conda create -n MulT python=3.6
conda activate MulT
conda install tensorflow-gpu==2.1
pip install tensorflo
build
er
s:
构建
transform
er
文件里面的模块,训练过程中用到的是VitBuild
er
这个类。
数据处理文件:
resize:将图片resize成256*256的大小
图片先需要转换成png格式,直接打开cmd,在该文件夹下输入ren *.jpg *.png就好。
一、
Auto
ml
是什么
机器学习
模型都是由工程师和科学家团队精心设计出来的。这种手动设计的过程是非常困难的,因为模型
组件
的搜索空间可以非常巨大——一个典型的10层网络模型能有1010种可能的网络。因此,设计网络的过程通常需要花费优秀的
机器学习
专家大量的时间和经验。
为了让设计
机器学习
模型的过程变得更加简单,谷歌一直在探索自动设计
机器学习
模型的方法。研...
A Volumetric
Transform
er
for Accurate
3D
Tumor Segmentation
用于精确三维肿瘤分割的体积
Transform
er
Published: 2021
Patt
er
n Recognition on Novemb
er
01, 2021
论文:https://arxiv.org/abs/2111.13300
代码:https://github.com/himashi92/VT-UNet
提出了一种用于
3D
医学
图像分割
的
Transform
er
架构。为体
3D
医学
数据增强 示例+代码
3D
医学
数据数据增强库数据可视化原始图片数据增强操作Resiz
eR
andomcorpPadNormalizeCropFlipRotateElastic
Transform
RandomRotate90GaussianNois
eR
andomGammaGridDropoutCutoutAbsRandom blur (torchio)随机数据增强
数据增强是深度训练过程中一个重要的步骤,2d的数据增强现在已经比较成熟,官方也有自己的数据增强函数。然而,
3d
数据增强的代码却不是很多,这里分析
MIC @ DKFZ的批处理生成器
batchgen
er
ators是我们在德国癌症研究中心(DKFZ)的
医学
图像计算部门开发的python软件包,可满足我们所有深度学习数据扩充的需求。 尚不完美,但我们认为它足以与社区共享。 如果
遇到
错误,请随时与我们联系或打开github问题。
如果使用它,请引用以下工作:
Isensee Fabian, J&au
ml
;g
er
Paul, Wass
er
thal Jakob, Zimm
er
er
David, Pet
er
sen Jens, Kohl Simon,
Schock Justus, Klein Andre, Roß Tobias, Wirk
er
t Sebastian, Neh
er
Pet
er
, Dinkelack
er
Stefan,
K&ou
ml
;hl
er
Gregor, Mai
er
-Hein Klaus (2020). batchgen
er
ators - a
### 回答1:
Transform
er
是一种基于自注意力机制的神经网络模型,可以应用于
医学
图像分割
任务中。
医学
图像分割
是指将
医学
图像中的不同组织或器官分离出来,以便进行更精确的诊断和治疗。
Transform
er
模型可以通过学习图像中不同区域之间的关系,自动识别出不同的组织和器官,并将其分割出来。这种方法相比传统的基于卷积神经网络的
图像分割
方法,具有更好的准确性和鲁棒性。
### 回答2:
Transform
er
是一种用于
自然语言处理
的深度学习模型,但它近年来也被应用于
医学
图像分割
,特别是在使用
3D
医学
图像上的分割任务中取得了很好的结果。相比于传统方法,使用
Transform
er
的方法更加简单,并且能够在一些
医学
图像分割
模型中取得更好的性能。
医学
图像分割
是一个重要的任务,它旨在对
医学
图像中的不同区域进行标记并分离开来。这对
医学
诊断、治疗和疾病研究非常重要。但是,这是一个具有挑战性的任务,因为
医学
图像通常由大量的像素组成,不同的图像有着复杂的拓扑结构。
Transform
er
在
医学
图像分割
中的应用可归结为两种:一种是直接将
Transform
er
作为卷积神经网络(CNN)的初始输入部分,提取图像特征;另一种则是使用
Transform
er
作为整个
图像分割
模型中的框架。
对于第一种应用情况,研究人员通常使用一种称为“自注意力机制”的技术,将
Transform
er
嵌入到CNN中。自注意力指的是对于输入的任意两个位置$a_i$和$a_j$,通过一对注意力权重$w_{ij}$来计算它们之间的相互作用。这种方法将图像的不同区域之间的关系考虑在内,能够有效地提取图像特征。
而对于第二种应用情况,研究人员通常使用称为“
Transform
er
-UNet”的结构,它基于UNet的框架,其中UNet作为Encod
er
作用,而
Transform
er
模型作为Decod
er
。这种方法将
Transform
er
的优秀性能和UNet的优秀性能相结合,能够更准确地对
医学
图像进行分割。
总体来说,
Transform
er
在
医学
图像分割
中的应用非常有前途,并且已经被证明比传统的方法更具优势。虽然目前的研究还有许多局限性,但是对于未来发展的前景非常乐观。
### 回答3:
Transform
er
是一种基于自注意力机制(self-attention)的神经网络模型,主要应用于
自然语言处理
等领域。但近年来,
Transform
er
在
医学
影像分析领域中也被广泛应用。
医学
图像分割
是一种重要的
医学
影像分析技术,它可以将医疗影像中的组织结构进行分割和提取,为医生诊断和治疗提供有力支持。然而,
医学
影像分割
任务面临着复杂背景、低对比度、噪声等多种问题,这也就需要一些先进的深度学习技术来实现准确地分割。
传统的
医学
图像分割
方法往往基于基于卷积神经网络 (CNN),但是 CNN 在处理
医学
图像时,由于其固定大小的卷积核并不能很好地捕捉全局上下文信息,导致分割效果不尽人意。而
Transform
er
通过自注意力机制,可以同时处理全局特征,能够有效捕捉全局上下文信息,进而提高分割效果。
基于
Transform
er
,近期研究者提出了一种基于
Transform
er
的
医学
影像分割
模型——TransUnet。该模型遵循了传统的编码器-解码器结构,在编码器中使用了
Transform
er
模块来捕捉全局上下文信息,解码器中则使用了 U-Net 结构来进行分割。此外,研究者还在模型训练过程中采用了 Dice Loss,并使用了 Mixup 技术和数据增强策略来进一步提高模型的分割性能。
目前,TransUnet 在公开数据集 BRATS2020 上取得了 SOTA 的效果,证明了
Transform
er
在
医学
影像分割
任务上的潜力。虽然
Transform
er
在
医学
影像分割
方面的应用还处于初期阶段,但是它无疑是未来
医学
影像分析领域中的重要发展方向之一。