🤩 WGCNA | 值得你深入学习的生信分析方法!~(网状分析-第五步-高级可视化)
1写在前面
前面我们用
WGCNA
分析完成了一系列的分析,聚类分割模块。🥰
随后进一步筛选,找到与我们感兴趣的表型或者临床特征相关的模块,而且进行了模块内部分析。😘
再然后是对感兴趣模块进行功能注释,了解模块的功能及涉及的潜在机制。🥳
本期主要是介绍一些可视化的方法,大家了解一下吧。🥰
2用到的包
rm(list = ls())
library(WGCNA)
library(dplyr)
3示例数据
load("FemaleLiver-01-dataInput.RData")
load("FemaleLiver-02-networkConstruction-auto.RData")
4计算基因数与样本数
计算一下基因数和样本数吧,后面会用到。🤓
nGenes <- ncol(datExpr)
nSamples <- nrow(datExpr)
5基因网络的可视化
5.1 计算TOM
我们再重新计算一下
TOM
吧,
power
是6,之前的教程介绍过如何计算。
dissTOM <- 1-TOMsimilarityFromExpr(datExpr, power = 6)

5.2 转换一下并可视化
这里我们需要转换一下
dissTOM
, 可以增强对比,方便进行可视化。
plotTOM <- dissTOM^6
diag(plotTOM) <- NA
sizeGrWindow(9,9)
TOMplot(plotTOM, geneTree, moduleColors, main = "Network heatmap plot, all genes")
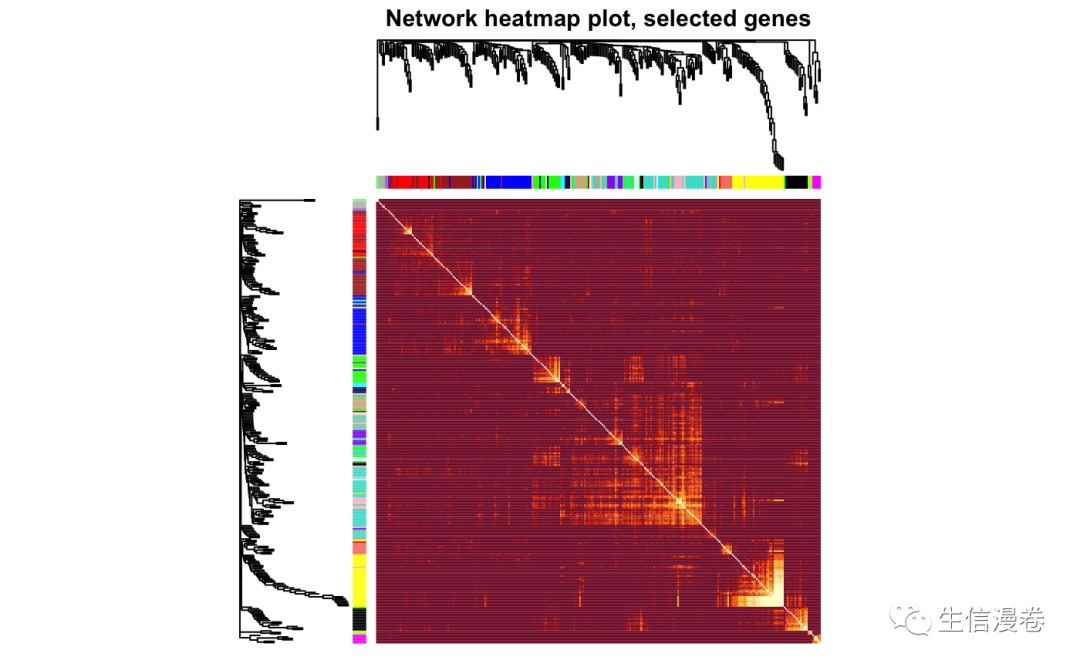
5.3 随机选择基因
由于数据量比较大,这里我们就随机选择
300
个基因来进行可视化吧。😉
nSelect <- 300
set.seed(123)
select <- sample(nGenes, size = nSelect)
selectTOM <- dissTOM[select, select]
selectTree <- hclust(as.dist(selectTOM), method = "average")
selectColors <- moduleColors[select]
5.4 可视化
来吧,展示!~😜
浅色代表
low adjacency (overlap)
,深色代表
higher adjacency (overlap)
。🤨
这里需要补充一下,如果你的
TOM
是用
blockwise
计算得到的,需要对每个
block
都运行一遍这个
code
,这里不做具体介绍了,写个循环吧。😏
sizeGrWindow(9,9)
plotDiss <- selectTOM^6
diag(plotDiss) <- NA
TOMplot(plotDiss, selectTree, selectColors, main = "Network heatmap plot, selected genes")
6网络的eigengenes可视化
6.1 计算模块的eigengenes
MEs <- moduleEigengenes(datExpr, moduleColors)$eigengenes
6.2 合并module与traits信息
weight <- as.data.frame(datTraits$weight_g)
names(weight) <- "weight"
MET <- orderMEs(cbind(MEs, weight))
6.3 可视化
sizeGrWindow(5,7.5)
par(cex = 0.9)