R语言中缺失值的识别与可视化
在问卷数据或实验数据中,经常会包含由于未作答、设备故障或误编码数据的缘故出现缺失值。处理带有缺失值的数据是一件很普遍的事,同时也是一件麻烦事,本文教大家如何在R中找出缺失值,探索缺失值模式和处理缺失值。选用R语言的原因是因为它可以将缺失值可视化,非常直观地看出缺失值在哪里。
数据缺失的原因
数据缺失有多种原因,如:
- 调查对象忘记回答一个或多个问题;
- 调查对象拒绝回答敏感问题;
- 感觉疲劳而没有完成一份很长的问卷;
- 错过了约定;
- 过早地从研究中退出;
- 记录设备出现问题;
- 网络连接失效;
- 数据误记等。
在R中,缺失值以符号NA表示,NA就是Not Available的缩写。本文主要使用VIM和mice包,如果你的R中没有安装过这两个包,需要先安装然后载入。
#安装VIM和mice包
install.packages(c("VIM","mice"))
# 载入VIM和mice包
library(VIM)
library(mice)本次分析使用的数据集sleep就是VIM包中包含的数据集,看这个名字就是这些数据和睡眠有关,其实是哺乳动物睡眠数据。来源于Allison和Chichetti (1976)的研究。
自变量:生态学变量、体质变量
因变量:睡眠变量
生态学变量包含:物种被捕食程度(Pred),睡眠时的暴露程度(Exp),面临的总危险度(Danger)。1(低)到5(高)计分。
体质变量包含:体重(BodyWgt,单位kg),脑重(BrainWgt,单位g),寿命(Span,单位年),妊娠期(Gest,单位为天)。
睡眠变量包含:做梦时长(Dream),不做梦时长(NonD)以及它们的和(Sleep)。
在分析缺失值之前,我们先熟悉一下sleep数据集的构成。
# 查看sleep数据框中的信息
str(sleep)
'data.frame': 62 obs. of 10 variables:
$ BodyWgt : num 6654 1 3.38 0.92 2547 ...
$ BrainWgt: num 5712 6.6 44.5 5.7 4603 ...
$ NonD : num NA 6.3 NA NA 2.1 9.1 15.8 5.2 10.9 8.3 ...
$ Dream : num NA 2 NA NA 1.8 0.7 3.9 1 3.6 1.4 ...
$ Sleep : num 3.3 8.3 12.5 16.5 3.9 9.8 19.7 6.2 14.5 9.7 ...
$ Span : num 38.6 4.5 14 NA 69 27 19 30.4 28 50 ...
$ Gest : num 645 42 60 25 624 180 35 392 63 230 ...
$ Pred : int 3 3 1 5 3 4 1 4 1 1 ...
$ Exp : int 5 1 1 2 5 4 1 5 2 1 ...
$ Danger : int 3 3 1 3 4 4 1 4 1 1 ...
# 我们也可以用head()查看数据集中的数据,默认给出前6行
head(sleep)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 3
2 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 3
3 3.385 44.5 NA NA 12.5 14.0 60 1 1 1
4 0.920 5.7 NA NA 16.5 NA 25 5 2 3
5 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 4
6 10.550 179.5 9.1 0.7 9.8 27.0 180 4 4 4我们可以看到sleep这个数据集是个数据框data.frame,包含10个变量,62条数据。其中前7个的数据类型为数值型num,后三个为整数型int。
比如第一条数据中,该动物的体重是6654千克,脑重是5712克,不做梦时长缺失,做梦时长缺失,睡眠时间3.3小时,寿命38.6年,妊娠期645天,被捕食程度3分,睡眠时的暴露程度5分,面临的总危险度3分。
识别缺失值
(1)is.na函数
在R中,可以用函数is.na( )检测缺失值是否存在。它将返回一个相同大小的对象,如果某个元素是缺失值,相应的位置将被改写为TRUE,不是缺失值的位置则为FALSE。
#检测sleep数据集的缺失值
is.na(sleep)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
[1,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
[2,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[3,] FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
[4,] FALSE FALSE TRUE TRUE FALSE TRUE FALSE FALSE FALSE FALSE
[5,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[6,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE由于篇幅限制,我们就看前6条数据,拿第一条数据举例,可以看出该动物在NonD和Dream上的数据是缺失的,在其他变量上没有缺失数据。
使用sum汇总有缺失值的数据条数,可以看到sleep数据集共有38条缺失值。
sum(is.na(sleep))
[1] 38如果想单独查看某个变量上共含几条缺失值,只需在数据集后加上变量名。这里我们查看有多少条数据在寿命这个变量上是缺失的。
sum(is.na(sleep$Span))
[1] 4(2)complete.cases函数
函数complete.cases( )可以用来识别矩阵或数据框中没有缺失值的行。若每行都包含完整实例,则返回TRUE的逻辑向量;若每行有一个或多个缺失值,则返回FALSE。
# 加载VIM包中的哺乳动物睡眠数据集sleep
data(sleep, package="VIM")
# 列出没有缺失值的行
sleep[complete.cases(sleep),]
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
2 1.000 6.60 6.3 2.0 8.3 4.5 42.0 3 1 3
5 2547.000 4603.00 2.1 1.8 3.9 69.0 624.0 3 5 4
6 10.550 179.50 9.1 0.7 9.8 27.0 180.0 4 4 4
7 0.023 0.30 15.8 3.9 19.7 19.0 35.0 1 1 1
8 160.000 169.00 5.2 1.0 6.2 30.4 392.0 4 5 4
9 3.300 25.60 10.9 3.6 14.5 28.0 63.0 1 2 1
10 52.160 440.00 8.3 1.4 9.7 50.0 230.0 1 1 1我们可以看到以上这些数据均没有缺失值。下面我们在语句前加入感叹号!,!代表“非”,所以sleep[!complete.cases(sleep),]的意思就是列出有缺失值的行。
# 列出有一个或多个缺失值的行
sleep[!complete.cases(sleep),]
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 3
3 3.385 44.5 NA NA 12.5 14.0 60 1 1 1
4 0.920 5.7 NA NA 16.5 NA 25 5 2 3
13 0.550 2.4 7.6 2.7 10.3 NA NA 2 1 2
14 187.100 419.0 NA NA 3.1 40.0 365 5 5 5
19 1.410 17.5 4.8 1.3 6.1 34.0 NA 1 2 1
20 60.000 81.0 12.0 6.1 18.1 7.0 NA 1 1 1从以上数据可以看出,每行至少有一个缺失值NA,也可能包含多个NA。
(3)md.pattern函数
mice包中的md.pattern,形成缺失表
md.pattern(data)
md.pattern(sleep)
BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD
42 1 1 1 1 1 1 1 1 1 1 0
2 1 1 1 1 1 1 0 1 1 1 1
3 1 1 1 1 1 1 1 0 1 1 1
9 1 1 1 1 1 1 1 1 0 0 2
2 1 1 1 1 1 0 1 1 1 0 2
1 1 1 1 1 1 1 0 0 1 1 2
2 1 1 1 1 1 0 1 1 0 0 3
1 1 1 1 1 1 1 0 1 0 0 3
0 0 0 0 0 4 4 4 12 14 381表示无缺失,0表示有缺失。
第一列表示该行模式数据的个数,该行没有缺失值,也就是在数据集sleep中,没有缺失值的数据共42条。缺失Span的数据共2条,同时缺失Dream和NonD的数据共9条。
(4)aggr函数
aggr是VIM包中的函数,可以形成缺失图。
aggr(sleep, prop = F, number = T)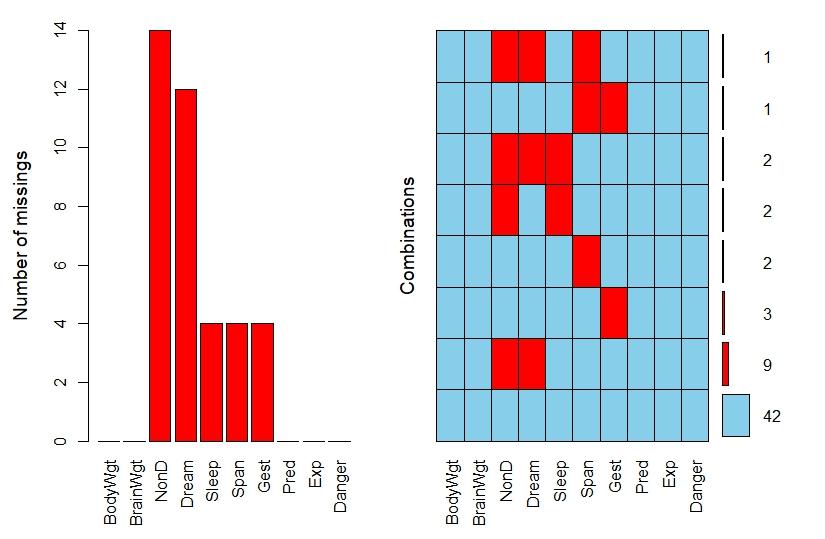
红色表示缺失的数量,这是因为prop是false。当prop是true的时候,纵轴便是缺失值的比例。
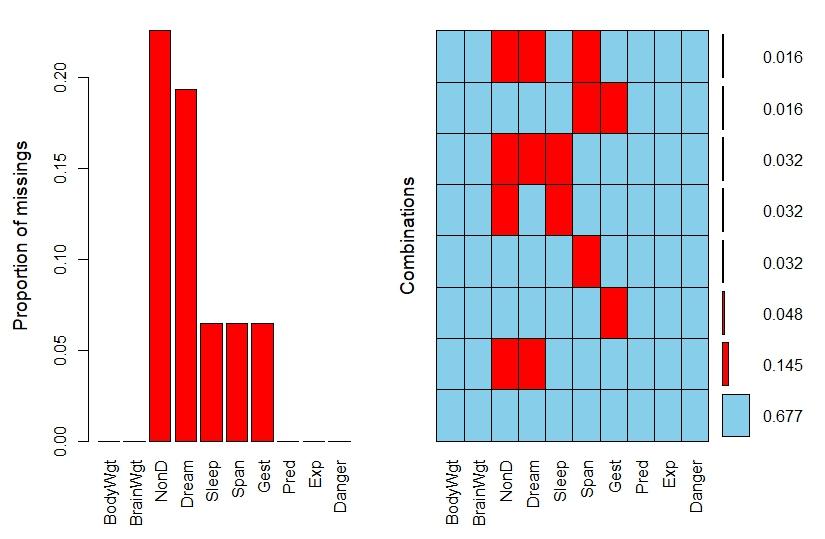
我们也可以将aggr的结果赋值给a,然后查看a
a <- aggr(sleep, P= F, N= T)
summary(a)
Missings per variable:
Variable Count
BodyWgt 0
BrainWgt 0
NonD 14
Dream 12
Sleep 4
Span 4
Gest 4
Pred 0
Exp 0
Danger 0
Missings in combinations of variables:
Combinations Count Percent
0:0:0:0:0:0:0:0:0:0 42 67.741935
0:0:0:0:0:0:1:0:0:0 3 4.838710
