单细胞工具箱|singleR-单细胞类型自动注释(含数据版)
本文首发自 “生信补给站” 单细胞工具箱|singleR-单细胞类型自动注释(含数据版)
单细胞研究中细胞类型注释是很重要的环节,大致分为人工注释和软件注释。
(1)人工注释需要借助文献检索marker或者结合常用的注释数据库-
cellMarker( http:// biocc.hrbmu.edu.cn/Cell Marker/ ),
PanglaoDB( https:// panglaodb.se/ ),
CancerSEA( http:// biocc.hrbmu.edu.cn/Canc erSEA/ )等,比较考验研究者的相关背景和精力,优点在于准确性相对较好。
(2)软件自动化注释一般是使用软件内置数据集进行注释,操作相对简单。但是准确性会相对稍差,不过可以作为一种很好的辅助注释手段。
自动化注释的软件很多,本次先简单的分享如何使用singleR进行自动注释。
SingleR是一个用于对单细胞RNA-seq测序(scRNA-seq)数据进行细胞类型自动注释的R包(Aran et al.2019)。依据已知类型标签的细胞样本作为参考数据集,对测试数据集中的细胞进行标记注释。
一 内置数据库
使用SingleR的最简单方法是使用内置参考对细胞进行注释。singleR自带的7个参考数据集,其中5个是人类数据,2个是小鼠的数据:
BlueprintEncodeData
Blueprint (Martens and Stunnenberg 2013) and Encode (The ENCODE Project Consortium 2012) (人)
DatabaseImmuneCellExpressionData
The Database for Immune Cell Expression(/eQTLs/Epigenomics)(Schmiedel et al. 2018)(人)
HumanPrimaryCellAtlasData
the Human Primary Cell Atlas (Mabbott et al. 2013)(人)
MonacoImmuneData
, Monaco Immune Cell Data - GSE107011 (Monaco et al. 2019)(人)
NovershternHematopoieticData
Novershtern Hematopoietic Cell Data - GSE24759(人)
ImmGenData
the murine ImmGen (Heng et al. 2008) (鼠)
MouseRNAseqData
a collection of mouse data sets downloaded from GEO (Benayoun et al. 2019).鼠)
二 数据库,R包
2.1 singleR包安装
#if (!requireNamespace("BiocManager", quietly = TRUE))
# install.packages("BiocManager")
BiocManager::install("SingleR")
BiocManager::install("Seurat")2.2 加载数据集,数据
加载数据库,可能会比较慢。建议下载好数据库后进行保存。
library(SingleR)
library(celldex)
library(Seurat)
library(pheatmap)
##下载注释数据库
hpca.se <- HumanPrimaryCellAtlasData()
hpca.se
#直接load下载好的数据库
load("HumanPrimaryCellAtlas_hpca.se_human.RData")
load("BlueprintEncode_bpe.se_human.RData")关注“生信补给站”后台回复“注释”即可获取注释数据库以及测试数据的Rdata文件。
2.3 查看seurat结果
使用 单细胞工具箱|Seurat官网标准流程 得到的pbmc的seurat的结果,
(1)查看seuret聚类结果
load("pbmc_tutorial.RData")
meta=pbmc@meta.data #pbmc的meta文件,包含了seurat的聚类结果
head(meta)
orig.ident nCount_RNA nFeature_RNA percent.mt percent.HB RNA_snn_res.0.5 seurat_clusters labels
AAACATACAACCAC-1 pbmc3k 2419 779 3.0177759 0 0 0 T_cells
AAACATTGAGCTAC-1 pbmc3k 4903 1352 3.7935958 0 3 3 B_cell
AAACATTGATCAGC-1 pbmc3k 3147 1129 0.8897363 0 2 2 T_cells
AAACCGTGCTTCCG-1 pbmc3k 2639 960 1.7430845 0 1 1 Monocyte
AAACCGTGTATGCG-1 pbmc3k 980 521 1.2244898 0 6 6 NK_cell
AAACGCACTGGTAC-1 pbmc3k 2163 781 1.6643551 0 2 2 T_cells
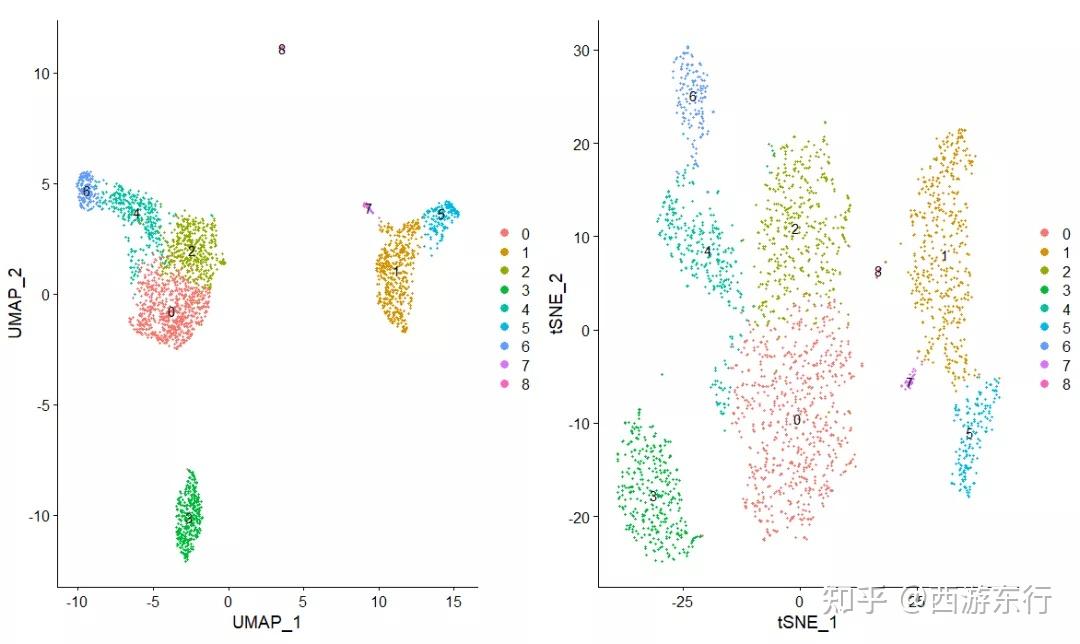
(2)查看umap和tsne图
plot1 <- DimPlot(pbmc, reduction = "umap", label = TRUE)
plot2<-DimPlot(pbmc, reduction = "tsne",
label = TRUE)
plot1 + plot2
三 singleR注释
3.1 singleR使用内置数据集注释
#进行singleR注释
pbmc_for_SingleR <- GetAssayData(pbmc, slot="data") ##获取标准化矩阵
pbmc.hesc <- SingleR(test = pbmc_for_SingleR, ref = hpca.se, labels = hpca.se$label.main) #
pbmc.hesc
#seurat 和 singleR的table表
table(pbmc.hesc$labels,meta$seurat_clusters)

3.2 绘制umap/tsne图
pbmc@meta.data$labels <-pbmc.hesc$labels
print(DimPlot(pbmc, group.by = c("seurat_clusters", "labels"),reduction = "umap"))

3.3 使用多个数据库注释
使用BP和HPCA两个数据库综合注释,使用list函数读入多个数据库
pbmc3 <- pbmc
pbmc3.hesc <- SingleR(test = pbmc_for_SingleR, ref = list(BP=bpe.se, HPCA=hpca.se),
labels = list(bpe.se$label.main, hpca.se$label.main))
table(pbmc3.hesc$labels,meta$seurat_clusters)
pbmc3@meta.data$labels <-pbmc3.hesc$labels
